资讯中心
作者:香雪精准医疗-郑红俊
发布时间:2020-03-11
阅读:14576
一场新肺炎在武汉肆虐,继而扩展到全国,甚至到了其他国家和地区。科学家们分析出这是一种新的冠状病毒在捣鬼。这不禁让人想起17年前的SARS冠状病毒,真是不寒而栗。大魔王又来了吗?这大过年的,还让不让人过年了?
很快,新冠病毒的基因组发表了,不同团队发表的序列高度一致,是同一种病毒没跑了(序列编号MN908947,NC_045512,MN938384,MN975262等等)。更多新序列可以在GISAID网站索取。
让我们来看看这害人的小妖精是何方妖孽。
首先,冠状病毒长这样,表面很多刺头被人们美化成皇冠上的明珠,故而得名冠状病毒,其实一个带刺的大球球,是不是有点像小时候玩的苍耳?苍耳很容易粘到衣服上,靠的就是表面密密麻麻的刺。冠状病毒也是一样,图中紫色的蛋白就是它的刺,特异性识别人体粘膜蛋白ACE2,所以保护好我们口鼻眼的粘膜就很重要了。
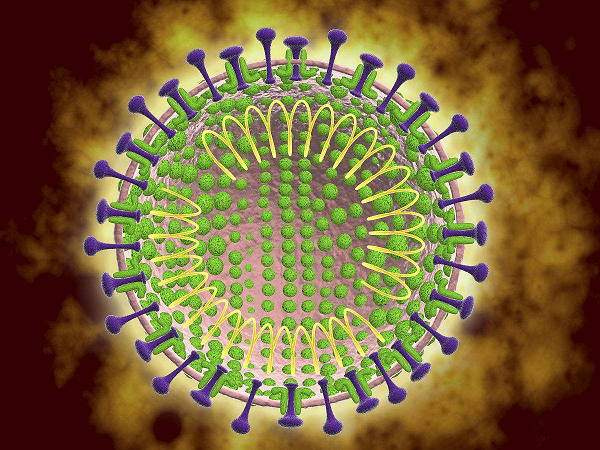
图1: 冠状病毒(图片来自百度)
于是就有科学家从这根刺上下手,想办法封闭掉这刺,就避免了感染人体,包括抗体,小RNA,疫苗,细胞免疫等技术。也有其他科学家团队从其他角度入手,此处不一一列举了。
我们今天主要是来认识这根刺(英文名SPIKE,缩写S)。
新冠病毒的S蛋白有1273个氨基酸,有发表的其他冠状病毒S蛋白序列稍长稍短,不一而足。总体而言,S蛋白是三聚体,俗称三花聚顶。它有一个长长的杆,和一个大大的头。杆的位置序列保守,如果针对这个部位用抗体封闭就举一反三了,但是杆子被大头挡住了,很难下手。大头成了绕不过去的一道坎,但是大头的序列就不如杆的序列保守了,它多变,像个傲娇的小女人。大头也叫RBD,就是和人体蛋白结合的区域,在不同的冠状病毒中序列不一样,也就导致和人体蛋白结合的强弱不一。很明显,新冠比SARS引起的病症要弱,毒性低,但是潜伏期长,传染更隐秘。这是病毒进化的结果,更好地依赖宿主生存和传播,而不是一上来就搞死宿主。
病毒究竟是怎样进化得更聪明的呢? 这是一个深奥的课题,病毒在地球存在了几十亿年,有150万种之多。人类才出现几十万年,我们认识到的病毒只是其中的冰山一角,所以我们要尊重它们的存在,这个自然界真正的霸主。
比较已经发布的病毒序列可以看出一点端倪。于是我们把大头的氨基酸序列丢到数据库中比对,终于发现了一点线索。图2展示的结果显示出与新冠病毒的大头最相似的序列是来自蝙蝠的SARS样冠状病毒(AGZ48806,ATO98157)。巧的是,这两条序列都是武汉病毒所的石正丽的团队发表的。所以就有了网上讨伐石教授泄露病毒的谣言。序列虽然相似,但是要进化到完全一样,需要几十年的时间。几十年前还没这个团队呢。
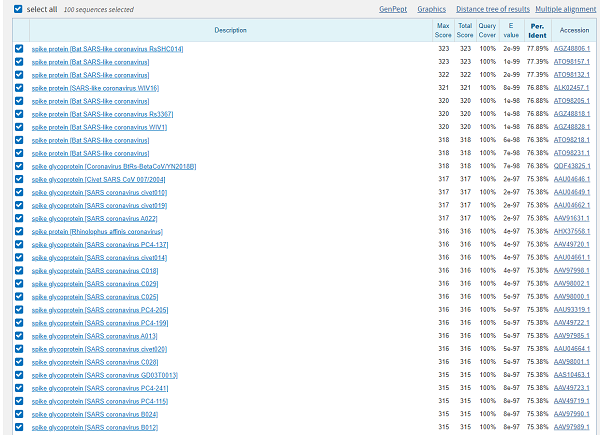
图2 大头的序列比对
好了,下面要划重点了。
当我们把最相似的几十条序列排好队仔细查看的时候,发现了一些关键氨基酸的区别。
EPI_ISL_410721 LCFTNVYADSFVVRGDEVRQIAPGQTGRIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNY
MN908947 LCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNY
ATO98157.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFLGCVLAWNTNSKDSSTSGNY
AGZ48806.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFLGCVLAWNTNSKDSSTSGNY
AGZ48828.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFTGCVLAWNTRNIDATQTGNY
AHX37558.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSSGNF
QDF43825.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
ATO98231.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
ATO98218.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV91631.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU04662.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU04649.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU04646.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
ABF68957.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV97986.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV97989.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV97990.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV49719.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV49723.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAS10463.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV98001.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU04664.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV97985.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV49722.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU93319.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV98000.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAU04661.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAV49720.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
AAS00003.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
BAE93401.1 LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
3SCI_E LCFSNVYADSFVVKGDDVRQIAPGQTGVIADYNYKLPDDFMGCVLAWNTRNIDATSTGNY
***:********::**:********** ************ ***:***:.. *:. **
图3 与大头最相似的序列的比对
序列相似性从上到下渐行渐远。最上面的一行序列是来自穿山甲的(EPI_ISL_410721),是最相似大头的(MN908947),所以穿山甲有可能是中间宿主。其次相似的是来自蝙蝠的冠状病毒样序列(ATO98157,AGZ48806),标黄的氨基酸N(天冬酰氨)和大头一致。其他序列均来自SARS,在这个关键氨基酸上却是R(精氨酸)。由此可见新冠与蝙蝠来源的更接近,而距离SARS远些。
凭什么说这个标黄的氨基酸N是关键氨基酸呢?
那是在很久很久以前啦,有人发表了SARS的刺头与人体蛋白ACE2的复合结构(PDB网站搜索2AJF),这个结构就展示了刺头上能结合ACE2的关键氨基酸,如图4所示。
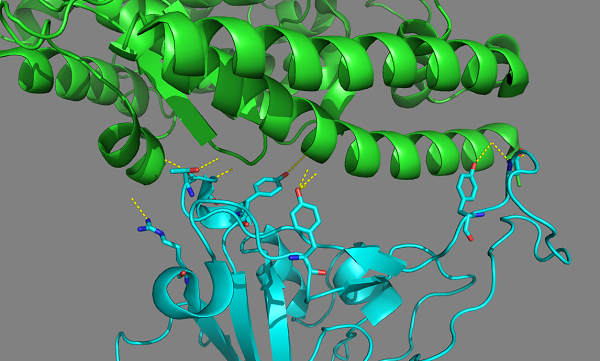
图4: SARS的刺头结合人体ACE2蛋白的结构展示(PDB:2AJF)
图中绿色的波浪卷是人体ACE2,青色的是SARS刺头蛋白,二者之间形成氢键(黄色虚线)的关键氨基酸侧链都在没有固定结构的环上。
那么新冠的大头又会怎样呢?
通过软件SWISS MODEL模拟新冠病毒大头的RBD结构,发现它与SARS刺头结构很相似,而上文提到的标黄的关键氨基酸N在图5中左边显示,明显比SARS刺头的R要短,不能与ACE2有效形成氢键,其他关键氨基酸不变,故而新冠大头的亲和力弱于SARS刺头。这也就解释了新冠毒性弱的根本原因。
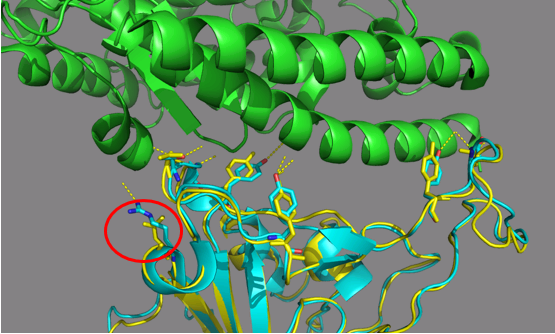
图5 模拟的新冠病毒大头与人体蛋白ACE2结合的结构
图中绿色的波浪卷是人体ACE2,青色的是SARS刺头蛋白,黄色是模拟的新冠的大头。红圈显示关键氨基酸的变化R变成N之后,侧链变短不能形成氢键。而其他氨基酸一致。
大头RBD中的结合区域的氨基酸序列如下:

大家留意到序列中几个氨基酸标红了,这些就是大头序列中与人体蛋白ACE2会结合的关键氨基酸。其中标黄的则是与SARS不一样的氨基酸,其他标红的一致。值得一提的是,近期发表的穿山甲中携带的冠状病毒序列与新冠病毒的关键氨基酸完全一致。
好了,重点讲完了。今天介绍了新冠给大家认识,从病毒总体到S蛋白,然后到大头区域。
从根本上认识病毒,才让我们不畏惧它,才能找到它的弱点战胜它。
参考:
2 PLoS Pathog. 2017 Nov 30; 13(11):e1006698. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus.
3 Nature. 2013 Nov 28; 503(7477): 535–538. Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor.
4 Viruses. 2019 Nov; 11(11): 979. Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)
5 Science. 2005 Sep 16;309(5742):1864-8. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor.