资讯中心
作者:香雪生命科学--卢华
发布时间:2021-04-29
阅读:15716
大家伙可能都听说过,在2003年经过多个国家的多年努力,完成了宏大的人类基因组计划,该工程通过测序确定了人类染色体中30亿个碱基对组成的DNA序列,被誉为生命科学研究的“登月计划”。
对整个基因组(生物体中所有的DNA)进行测序是一项复杂的任务。它需要将基因组DNA先分解成许多较小的片段,然后对这些小片段进行测序,接着将这些序列组装成完整的DNA序列。 随着技术不断发展和改进,目前的全基因组测序速度更快,成本更低。本文给大家介绍经典的测序方法——Sanger测序,然后简要介绍新一代测序技术
Sanger测序:链终止法
Sanger测序技术是由英国生物化学专家Sanger教授和其同事于1977年开发,该方法测定的DNA片段一般不超过900个碱基。在人类基因组计划中,Sanger测序用于确定人类DNA中许多相对较小片段的序列,但这些片段不一定小于900 个碱基,研究人员可以利用多轮Sanger测序沿着每个片段进行测序,再根据重叠部分对片段进行比对,组装较大DNA区域的序列,最终组装整个染色体序列。
Sanger测序原理
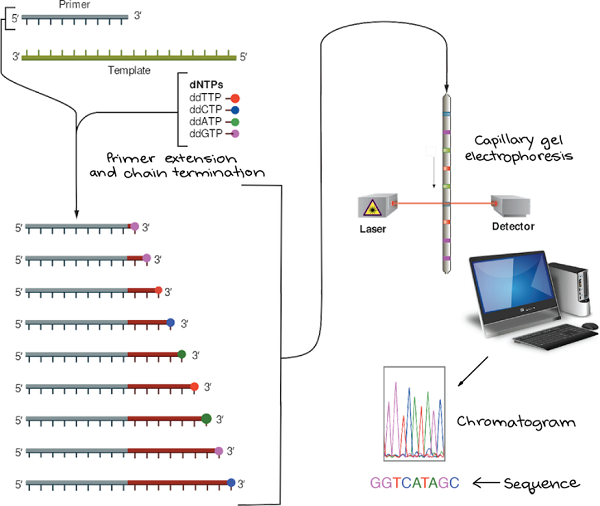
Sanger测序涉及制备靶DNA区域的多个拷贝,它的组成类似于生物体中复制DNA或体外复制DNA的聚合酶链反应(PCR)所需的成分:DNA聚合酶,引物,四种脱氧核苷酸(dNTP)以及待测序的DNA模板。
当然,Sanger测序反应也有其独特的成分,这也是其核心原理:四种不同染料标记的双脱氧核苷酸(ddNTP)。相比常规的脱氧核苷酸,双脱氧核苷酸糖环的3’碳原子上缺少羟基。因此,在DNA合成过程中一旦双脱氧核苷酸添加到链中,将无法正常形成磷酸二酯键,进而使DNA合成反应终止。该DNA链也就以染料标记的双脱氧核苷酸结尾,可以利用凝胶电泳和显影技术根据条带位置确定待测DNA片段的序列。

图一 Sanger测序
Sanger测序基本过程
在DNA合成反应体系中加入一定比例带有染料标记的ddNTP,其数量要比普通核苷酸少得多。反应完成后,体系中包含了不同长度的DNA片段,终止于待测DNA序列的每个核苷酸位置,并且片段的末端被带有特定染料的ddNTP标记。
然后,将片段通过一根含有凝胶基质的毛细管,该过程也称为凝胶电泳。短片段能够快速地穿过凝胶,而长片段相对移动较慢。当每个片段越过管子末端时,都会被激发光照射,从而检测出ddNTP上附着的染料。
图二 测序流程
最小的片段(在引物后仅插入了一个核苷酸)首先穿过终点线,然后是第二个最小的片段(在引物后插入了两个核苷酸),依此类推。因此,检测器记录的数据由一系列荧光强度峰组成,可以根据检测器上一个接一个记录的染料颜色,从色谱图中读取待测DNA的序列。
用途和局限性
Sanger测序可为较长的DNA片段(最多约900个碱基对)提供高质量的测定,主要用于对单个DNA片段进行测序,如细菌生产的质粒或者是PCR克隆的DNA。
但是,对于大型项目,如整个基因组或宏基因组(微生物菌群的“集体基因组”)测序,Sanger测序昂贵且效率低下。对于这类任务,新的高通量测序技术更快,更便宜。
Next-generation sequencing
最新的DNA测序技术统称为下一代测序(NGS),也称高通量测序。有许多使用不同技术的新一代测序方法,但大多数具有相同的特点。相比传统的Sanger测序,NGS支持多个测序反应同时进行,测序反应体系小,可以在芯片上一次完成多组反应等优点。但是,其测序长度相对要短一些,通常为50-700个碱基。
从概念上讲,下一代测序有点像同时运行大量微小的Sanger测序反应。由于这种并行化和小规模,与Sanger测序相比,新一代测序技术可以更快,更便宜地对大量DNA进行测序。例如,在2001年,对人类基因组进行测序的成本接近1亿美元。但到了2015年只需要大约1000美元!
为什么快速廉价的测序很重要?对DNA序列进行常规测序的能力为生物学研究和生物医学应用开辟了新的可能性。例如,低成本的测序是朝着精准医学迈出的一步,也就是说,根据他或她基因组中的变异基因,可以针对个体差异量身定制个性化医疗方案。
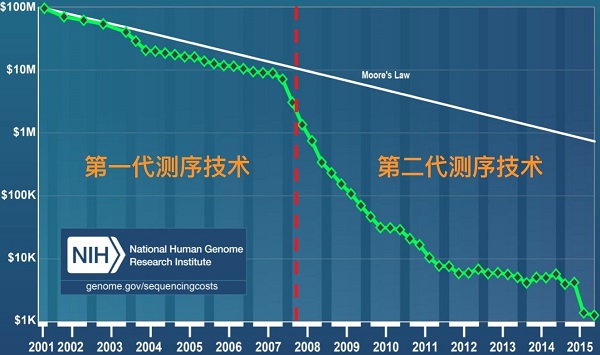
图三 测序成本比较
目前,第二代高通量测序技术以Roche公司的454平台、ABI公司的SOLiD平台和illumina公司的Solexa平台为代表。454平台采用了焦磷酸测序法,是首个被发明的商用二代测序仪。SOLiD平台是由独特的连接酶测序法发展而来,检测过程中两个碱基确定一个荧光信号,相当于一次能检测两个碱基,因此该方法也被称为两碱基测序法。Solexa平台是现在应用最广泛的二代测序平台,作为测序界的巨鳄,占据了全球70%以上的测序仪器市场份额。其测序原理是基于可逆性链终止物和桥式PCR等专有技术的边合成边测序法(SBS)。
第二代测序技术常用的应用包括小型全基因组测序(微生物和病毒)、靶向基因测序(基于扩增子、基因panel)、转录组测序(总RNA-Seq、mRNA-Seq和基因表达图谱分析)、甲基化测序以及16S宏基因组测序等。
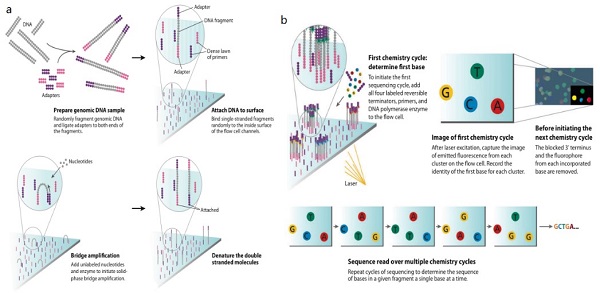
图四 SBS流程
近几年,随着关键性技术的突破和革命性的发明,诞生了第三代高通量测序技术。第三代测序技术也称为单分子测序技术,根据测序技术原理主要分为两大阵营:第一阵营是单分子荧光测序技术,代表性技术为Pac Bio公司的SMRT技术。该技术为了实现实时测序,利用零模波导孔技术避免了反应体系中游离的荧光标记核苷酸产生的强大荧光背景,同时开发出新的核苷酸荧光标记方法,将传统的碱基荧光标记改为标记磷酸基团,使得标记物会在DNA合成过程中自动脱落,大大提高了DNA聚合酶的活性和测序读长。第二阵营为Oxford Nanopore Technologies公司的单分子纳米孔测序技术。与以往的光信号检测手段不同,该方法采用电信号检测,不依赖DNA聚合酶,因此理论上能够做到超长的测序读长。
相比第二代测序,第三代测序最大特点是单分子测序,过程中不涉及PCR过程,因此能够有效避免PCR扩增的偏向性和引入突变,缺失等系统性错误。另外,第三代测序还能直接对DNA进行甲基化测序分析,不需要事先对基因组进行Bisulfite处理。但是,目前第三代测序平台仍存在一些不足,需进一步改进和完善,如测序成本和错误率较高等。

图五 MinlON测序仪
单细胞测序
自2013年,《Nature Methods》将单细胞测序评为年度技术,单细胞测序逐渐成为热点技术。为什么呢?一方面是因为测序成本大大降低,使得NGS已经成为实验室里的家常便饭。另一方面,细胞作为生命结构和功能的基本单位,以往的测序样品往往是组织样本或细胞群,检测结果是多个细胞的平均值,很容易掩盖了异质性细胞的信息。单细胞水平测序能够使测序结果更加全面,多层次,复杂程度更高。
简单来说,单细胞测序就是在测序前增加一步单细胞分离和标记的过程,常用的分离技术包括激光捕获显微切割、流式细胞分选、微孔捕获技术和微流控技术等。目前,单细胞测序主要应用于空间基因表达、单细胞染色质转座酶可接近性分析(ATAC)、单细胞免疫分析和单细胞基因表达等。通过以上应用我们可以探索细胞间异质性,鉴定新的靶点和生物标志物,分析配对的全长B细胞或T细胞受体、抗原特异性、细胞表面蛋白表达和染色质开放性等。
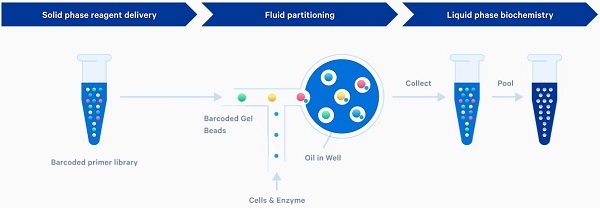
图六 10× Genomics的Chromium技术
小结
目前,NGS已经被广泛的应用于临床医学和基础研究中,作为破解生命遗传信息的重要手段,测序技术还在飞快的发展进化。相信在未来,测序技术能够帮助我们更全面的,更深层次的理解生命中的各种奥秘和疾病,甚至帮助我们攻克癌症。
参考文献:
1. Lewis, T. (2013, April 14) Human genome project marks 10th anniversary. In LiveScience.
2. National Human Genome Research Institute. (2016) DNA sequencing costs. In Large-scale genome sequencing and analysis centers (LSAC).
3. Obenrader, S. (2003) The Sanger method. In Sarah Obenrader: Molecular biology.
4. Reece, J.B., Urry, L.A., and Cain, M.L. (2011) Dideoxy chain termination method for sequencing DNA. In Campbell biology (10th ed., p. 410) San Francisco, CA: Pearson.
5. Thermo Fisher Scientific. (2016) Sanger sequencing method. In Sanger sequencing.
6. Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008;26(10):1135-1145. doi:10.1038/nbt1486
7. Zhang Z, Shen J, Wang H, et al. Effects of Graphene Nanopore Geometry on DNA Sequencing. J Phys Chem Lett. 2014;5(9):1602-1607. doi:10.1021/jz500498c
8. Mardis ER. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 2008;9:387-402. doi:10.1146/annurev.genom.9.081307.164359
9. Metzker ML. Sequencing technologies - the next generation. Nat Rev Genet. 2010; 11(1):31-46. doi:10.1038/nrg2626
10. Niedringhaus TP, Milanova D, Kerby MB, Snyder MP, Barron AE. Landscape of next-generation sequencing technologies. Anal Chem. 2011;83(12):4327-4341. doi:10.1021/ ac2010857
11. Method of the year 2013. Nat Methods. 2014;11(1):1. doi:10.1038/nmeth.2801